Vieillissement : Les mécanismes moléculaires de l’altération épigénétique
L’épigénétique, c’est l’étude des mécanismes qui agissent sur le génome pour contrôler l’expression des gènes. Moduler l’expression des gènes est important pour le maintien de l’équilibre cellulaire. Des altérations épigénétiques entraînent des changements dans l’expression des gènes, ce qui peut, à terme, dérégler les fonctions cellulaires. Ces modifications sont reconnues comme étant une des causes du vieillissement et sont retrouvées dans beaucoup de pathologies humaines.
Maîtriser l’expression des gènes se fait, en grande partie, en contrôlant l’état de condensation de la chromatine. Très condensée (hétérochromatine), elle ne permettra pas l’expression de gènes alors qu’une chromatine peu condensée (euchromatine) le permettra. Ces changements d’état se font par trois mécanismes : la méthylation de l’ADN, les modifications d’histones et le remodelage chromatinien. Avec l’âge, ces mécanismes sont bouleversés, influençant la longévité et le développement de maladies liées à l’âge.
Épigénétique : La méthylation de l’ADN
Les aberrations de méthylation de l’ADN sont les altérations épigénétiques les plus courantes et les plus étudiées. Avec le temps, des anomalies de méthylation s’accumulent et contribuent à la dérive épigénétique pendant le vieillissement. La méthylation de l’ADN, c’est l’ajout d’un groupement chimique méthyle (-CH3) sur une base cytosine (C) d’un dinucléotide CpG (une base cytosine suivit d’une base guanine et liées entre-elles par une liaison phosphate). Un grand nombre de ces dinucléotides CpG sont regroupés au niveau de régions génomiques appelées îlots CpG (séquences nucléotidiques avec une grande concentration de dinucléotides CpG) et environ la moitié de ces îlots CpG se trouve au niveau de régions régulatrices de gènes [1]. On comprend ainsi facilement que la méthylation joue un rôle important dans le contrôle de l’expression génique.
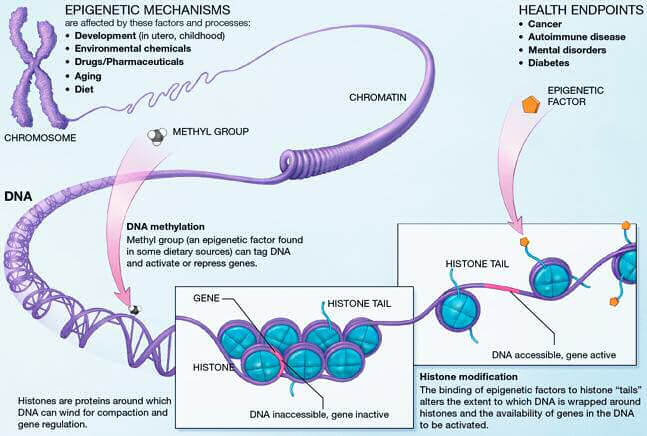
Des études approfondies des niveaux de méthylation des îlots CpG ont permis de mettre en évidence qu’il existe des régions différemment méthylées avec l’âge [1]. Certains de ces sites spécifiques sont associés au vieillissement et l’étude de leurs niveaux de méthylation permet de définir l’âge biologique de nos cellules : c’est l’horloge épigénétique. La méthylation de l’ADN a ainsi été développée comme biomarqueur performant et juste de prédiction de l’âge biologique [2].
Lors du vieillissement, on observe une perte globale du niveau de méthylation de l’ADN sur tout le génome (hypométhylation), mais certaines régions sont plus enclines à l’augmentation du niveau de méthylation (hyperméthylation). La méthylation de l’ADN est liée à la répression génique puisqu’elle induit la condensation de la chromatine, bloquant l’accès de la machinerie transcriptionnelle à l’ADN. Cette répression permet d’empêcher l’expression d’oncogènes (gènes qui favorisent le développement de cancers). Mais avec l’hypométhylation liée à l’âge, ces oncogènes peuvent être exprimés plus abondamment et participer au développement de cancers. Inversement, l’hyperméthylation touche certaines régions régulatrices de gènes en temps normal peu méthylées [3,4], c’est notamment le cas pour les gènes suppresseurs de tumeur. Une hyperméthylation sur ces gènes accentue aussi le risque de développer des cancers. Les changements de méthylation sont donc volontiers associés au développement de maladies, en particulier des cancers.
Épigénétique : Les modifications d’histones
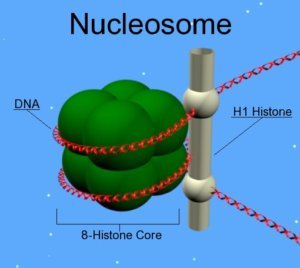
Les histones sont les protéines constitutives des nucléosomes, des complexes autour desquels l’ADN s’enroule pour former la chromatine. Elles subissent des modifications qui vont changer l’état de compaction de la chromatine, contrôlant ainsi l’expression des gènes. Ces modifications post-traductionnelles sont additionnées ou soustraites par des enzymes. Pendant le vieillissement, l’abondance et l’activité de ces enzymes régressent, entraînant des altérations de l’épigénome. A terme, cela provoque la perte de l’équilibre génique et favorise le développement de maladies liées à l’âge [3].
Les modifications les plus courantes qui touchent les histones sont les acétylations et les méthylations sur des résidus lysines. Des aberrations de ces modifications ont été observées chez les organismes modèles âgés et chez les personnes atteintes de progéria, une maladie rare engendrant un phénotype de vieillissement accéléré [5].
Il a été montré qu’une baisse de l’acétylation globale des histones joue un rôle important dans le vieillissement du cerveau de la souris et entraînerait une perte de la mémoire [6]. Dans le même sens, un niveau élevé de cette acétylation est associé à la longévité. De plus, des changements de méthylation sur les histones H3 sont associés à la perte de la structure de l’hétérochromatine, reconnue comme étant une marque du vieillissement. Les modifications de méthylation des histones sont associées au développement de cancers [4]. En plus de l’impact direct des aberrations de méthylation et d’acétylation d’histones sur le vieillissement, la méthylation des histones impacte aussi des voies impliquées dans le vieillissement, comme l’autophagie, la réponse aux stress environnementaux et la sénescence cellulaire.
Avec le temps, les histones subissent d’autres phénomènes. On observe une perte globale de leur nombre dans le génome avec l’âge. Il existe également des variants d’histones, cruciaux pour la régulation de la dynamique chromatinienne et retrouvés dans certaines conditions cellulaires. Le variant H3.3 de l’histone H3 est la forme qu’on retrouve majoritairement dans la chromatine des cellules sénescentes, et un excès de ce variant dans une cellule entraîne la sénescence cellulaire. Un autre variant lié à l’âge et également associé à la sénescence est le variant macroH2A de l’histone H2A, dont le niveau augmente avec l’âge. [6,7]
Épigénétique : Le remodelage chromatinien
En plus de la méthylation d’ADN et des modifications d’histones, d’autres mécanismes moléculaires influencent la structure de la chromatine par le biais de protéines spécifiques. C’est le cas des protéines HP1 (Heterochromatin protein 1) qui contribuent à former l’hétérochromatine. Lors du vieillissement, la quantité de protéines HP1 diminue, ce qui à terme entraîne la diminution des niveaux d’hétérochromatine, reconnus comme étant une marque de vieillissement. La perte d’hétérochromatine qui accompagne le vieillissement entraîne des changements de l’architecture globale du noyau et de l’expression des gènes, causant directement ou indirectement le vieillissement et la sénescence [6].
Les protéines HP1 permettent notamment de maintenir le lien entre l’hétérochromatine périphérique et la membrane nucléaire interne. La perte de ce lien suite à la baisse du niveau des protéines HP1 entraîne une perturbation chromatinienne et une régulation épigénétique défectueuse. Ce phénotype est retrouvé chez les patients atteints du syndrome de Huntchinson-Gilford (progéria) [8].
Par ailleurs, le complexe protéique NuRD (Nucleosome Remodelling Deacetylase) régule l’expression de gènes au moyen de deux activités enzymatiques : le remodelage des nucléosomes grâce à l’ATP (l’ATP est l’énergie de la cellule) et une activité déacétylase (le groupement acétyl est retiré) sur les histones. En raison des nombreuses sous-unités qui composent l’ensemble protéique et de leur hétérogénéité (multiples isoformes), les effets de NuRD sont complexes et parfois antagonistes. C’est le cas dans les cancers, où en fonction du tissu et du type de sous-unité, NuRD aura des incidences différentes sur la maladie [9]. Les études actuellement menées à son propos permettront de mieux comprendre son rôle dans les autres maladies liées au vieillissement.
Le vieillissement cellulaire peut de même s’accompagner de dysfonctionnement des protéines du groupe Polycomb. C’est un groupe de répresseurs transcriptionnels qui ciblent, entre autres, des gènes codant pour des facteurs de transcription, des protéines de signalisation et des régulateurs de toutes les voies majeures du développement. Une activité inappropriée de ces protéines joue un rôle dans la carcinogenèse, le développement et la progression de cancer [10,11].
Vers l’épigénétique du vieillissement
Les altérations épigénétiques sont largement étudiées depuis que ces mécanismes ont clairement été définis comme une des causes du vieillissement. A l’heure actuelle, des génomes entiers ont été étudiés pour connaître l’épigénome, c’est-à-dire l’ensemble des modifications épigénétiques de la cellule. L’épigénomique permet de comprendre l’impact de l’épigénétique sur le vieillissement et ouvre des possibilités de recherche et de traitement pour appréhender le processus de vieillissement.
Tout notre dossier sur l’épigénétique du vieillissement et la longévité :
Les altérations épigénétiques comme causes du vieillissement
Comprendre les altérations épigénétiques qui entraînent le vieillissement : récapitulatif des mécanismes impliqués.
Partie 2 : Épigénétique et ARNs non codants : le rôle des microARN et LncARN sur la longévité
La totalité de l’ADN ne code pas forcément des protéines ; les ARN non-codants tels que les microARN jouent aussi un rôle dans le processus de vieillissement.
Partie 4 : Epigenome editing, une technologie anti-vieillissement et pro-longévité
 Avec la naissance de l’épigénétique, une nouvelle technologie a vu le jour : l’epigenome editing, grâce à des avancées telles que l’outil CRISPR-Cas9.
Avec la naissance de l’épigénétique, une nouvelle technologie a vu le jour : l’epigenome editing, grâce à des avancées telles que l’outil CRISPR-Cas9.
Partie 1 : Les mécanismes moléculaires des altérations épigénétiques dans le vieillissement
Qu’est-ce que l’épigénétique ? Comment les altérations moléculaires entraînant le vieillissement en arrivent-elles à se produire ?
Partie 3 : L’horloge épigénétique, puissant outil de mesure du vieillissement
 On peut utiliser l’épigénétique comme un outil de mesure du vieillissement, en cartographiant les modifications qui surviennent avec l’âge.
On peut utiliser l’épigénétique comme un outil de mesure du vieillissement, en cartographiant les modifications qui surviennent avec l’âge.
Références :
[1] Yuanyuan Li and Trygve O Tollefsbol. Age-related epigenetic drift and phenotypic plasticity loss: implications in prevention of age-related human diseases. Epigenomics (2016) 8(12), 1637–1651.
[2] Steve Horvath. DNA methylation age of human tissues and cell types. Genome Biology, 14:R115.
[3] Andrey Tvardovskiy, Veit Schwammle, Stefan J. Kempf, Adelina Rogowska-Wrzesinska and Ole N. Jensen. Accumulation of histone variant H3.3 with age is associated with profound changes in the histone methylation landscape. Nucleic Acids Research, 2017, Vol. 45, No. 16, 9272–9289.
[4] Eric L. Greer and Yang Shi. Histone methylation: a dynamic mark in health, disease and inheritance, Nature Reviews, Genetics, Vol. 13 (May 2012), 343-357.
[5] Brenna S. McCauley and Weiwei Dang, Histone methylation and aging: Lessons learned from model systems, Biochim Biophys Acta. 2014 December ; 1839(12): 1454–1462. doi:10.1016/j.bbagrm.2014.05.008.
[6] Pal and Tyler Sci. Adv. 2016; 2 : e1600584
[7] Oriana Lo Re and Manlio Vinciguerra, Histone MacroH2A1: A Chromatin Point of Intersection between Fasting, Senescence and Cellular Regeneration, Genes 2017, 8, 367; doi:10.3390/genes8120367.
[8] Jia Liu, Xianhui Yin, Baohua Liu, Huiling Zheng, Guangqian Zhou, Liyun Gong, Meng Li, Xueqin Li, Youya Wang, Jingyi Hu, Vaidehi Krishnan, Zhongjun Zhou & Zimei Wang (2014) HP1α mediates defective heterochromatin repair and accelerates senescence in Zmpste24-deficient cells, Cell Cycle, 13:8, 1237-1247, DOI: 10.4161/cc.28105
[9] Jeannine Basta and Michael Rauchman. The Nucleosome Remodeling and Deacetylase (NuRD) Complex in Development and Disease, Transl Res. 2015 January ; 165(1): 36–47. doi:10.1016/j.trsl.2014.05.003.
[10] Wei Wang, Jiang-Jiang Qin, Sukesh Voruganti, Subhasree Nag, Jianwei Zhou and Ruiwen Zhang. Polycomb Group (PcG) Proteins and Human Cancers: Multifaceted Functions and Therapeutic Implications, Med Res Rev. 2015 November ; 35(6): 1220–1267. doi:10.1002/med.21358.
[11] Neil P. Blackledge, Nathan R. Rose, and Robert J. Klose. Targeting polycomb systems to regulate gene expression: modifications to a complex story, Nat Rev Mol Cell Biol . 2015 November ; 16(11): 643–649. doi:10.1038/nrm4067.
Anne Fischer

Author
Auteur
Anne is studying medicine science at the Institute of Pharmaceutical and Biological Science in Lyon and she has graduated with a Bachelor’s degree in molecular and cellular biology at the University of Strasbourg.
More about the Long Long Life team
Anne étudie les sciences du médicament à l’Institut des Sciences Pharmaceutiques et Biologiques de Lyon. Elle est titulaire d’une licence en biologie moléculaire et cellulaire de l’Université de Strasbourg.
En savoir plus sur l’équipe de Long Long Life
Dr Guilhem Velvé Casquillas

Author/Reviewer
Auteur/Relecteur
Physics PhD, CEO NBIC Valley, CEO Long Long Life, CEO Elvesys Microfluidic Innovation Center
More about the Long Long Life team
Docteur en physique, CEO NBIC Valley, CEO Long Long Life, CEO Elvesys Microfluidic Innovation Center
En savoir plus sur l’équipe de Long Long Life