La méthylation de l’ADN : un régulateur épigénétique puissant
Les modifications que subit notre ADN au cours de notre vie, en lien avec notre alimentation, notre environnement, des pathologies ou des phénomènes adaptatifs, que l’on appelle les modifications épigénétiques, sont intrinsèquement liées au vieillissement. L’avancée des méthodes quantitatives permettant des analyses complètes du génome (genome-wide associated studies ou GWAS) ont permis de mettre en lumière ces événements et d’étudier leur dérégulation.
Les modifications épigénétiques sont variées et peuvent intervenir à n’importe quel moment de notre vie en réponse à des signaux reçus par nos cellules : stress, tabagisme, alimentation, réponse hormonale… Elles peuvent être transitoires, pour faire face à une situation d’urgence spécifique, ou être pérennes, impactant ainsi directement l’expression des gènes et l’intégrité de notre ADN sur le long terme. Ces modifications peuvent prendre plusieurs formes et sont le fruit du travail d’enzymes spécifiques qui vont ajouter à notre ADN des petites structures, soit directement, soit sur les protéines associées à l’ADN comme les histones. Les modifications que l’ont connait le mieux à ce jour sont aussi les plus fréquentes : glycosylation (ajout de sucre), méthylation (ajout de groupes méthyl sur notre ADN), méthylation et acétylation des histones.
Elles ont un impact direct sur la transcription des gènes (et la génération d’une protéine complète) car elles peuvent bloquer des sites important sur l’ADN, empêchant l’accès à des séquences indispensables à la transcription. La méthylation peut ainsi conduire à l’inactivation d’un gène donné. Les altérations de la méthylation liées à l’âge sont référencées sous le nom de « dérive épigénétique » correspondant à un ensemble d’événements conduisant à la dé-méthylation graduelle du génome et l’hyper-méthylation de certaines zones, appelées îlots CpG [1].
Epigénétique et maladies
Des anomalies épigénétiques peuvent contribuer au développement de maladies. Cela a été prouvé pour les cancers [2] et, plus récemment, dans les maladies neurodégénératives [3, 4, 5] et les maladies métaboliques comme le diabète [6].
Les processus de méthylation, notamment, interviennent dans la régulation de nombreux événements, tels que la différenciation, la division, la multiplication ou la survie cellulaire. Ces phénomènes permettent en effet de réguler l’expression de certains gènes et, lorsque ces changements commencent à dysfonctionner, peuvent activer certains gènes qui ne devraient pas s’exprimer (comme les oncogènes) ou inhiber d’autres gènes qui auraient du rester actifs (comme les suppresseurs de tumeurs).
Méthylation et vieillissement
La méthylation est un type de modification épigénétique spécifique: elle a lieu au niveau des cytosines de notre ADN, une des quatre bases qui le forme (avec l’adénine, la guanine et la thymine). Une famille d’enzymes, les DNA methyltransferases (DNMTs), va permettre l’ajout d’un groupement méthyl (de formule chimique CH3) sur un des carbones qui forment la structure de la cytosine [7, 8]. Cette méthylation a un rôle majeur lors de l’embryogenèse puis, chez l’enfant et l’adulte, dans la régulation génétique. Elle est, par exemple, responsable de l’expression sélective de gènes maternels ou paternels, un mécanisme connu sous le nom « d’empreinte génétique » (genomic imprinting) qui permet notamment l’inactivation d’un des deux chromosomes X chez l’embryon fille. Après cette phase développementale, la méthylation va avoir lieu dans environ 1% des cytosines de l’ADN, le plus fréquemment au sein des îlots CpG [9].
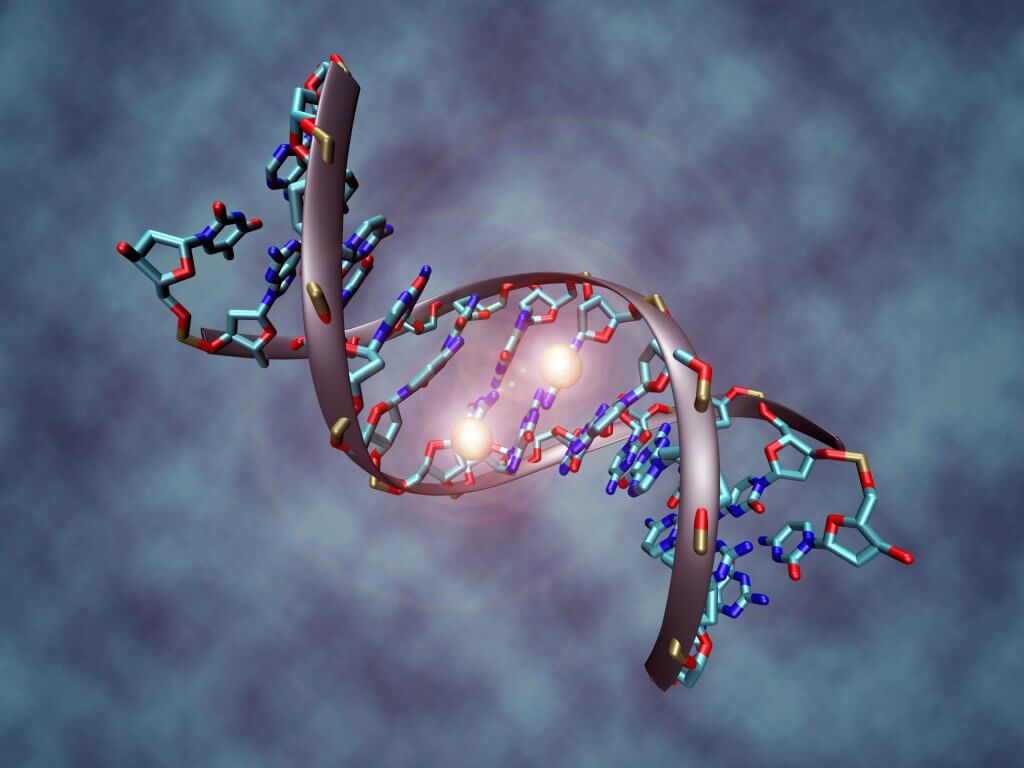
Lors du vieillissement, les DNMT commencent à méthyler de plus en plus certaines régions riches en cytosines et primordiales au fonctionnement de la transcription des gènes : les îlots CpG des promoteurs. Ce sont des zones non codantes (qui ne contiennent pas d’information génétique) situées en amont du gène et sur lesquelles vont se fixer les ARN polymérases, une famille d’enzymes responsable de la transcription. Lorsque ces promoteurs sont hyper-méthylés, les ARN polymérases ne peuvent plus se fixer et la transcription des gènes est inhibée [1, 8, 10].
Il existe donc un lien entre régulation génétique, méthylation et vieillissement. Plusieurs études ont d’ailleurs démontré que le taux de 5-méthyl-cytosine (5mC, la cytosine méthylée sur son cinquième carbone) était corrélé à l’âge [11, 12], donnant ainsi des outils supplémentaires à la métrologie du vieillissement.
Mesurer la méthylation, une information sur l’age biologique ?
Il existe des outils similaires à ceux utilisés pour le décryptage de notre génome permettant d’obtenir une information complète concernant les différentes modifications épigénétiques de notre ADN; on parle alors d’épigénome.
La méthylation peut être mesurée par ces techniques, comme l’Illumina (Infinium HumanMethylation BeadChip assays), un processus permettant d’obtenir, sur un génome entier, une information complète sur le nombre d’îlots CpG présents et combien ont été méthylés.
Une équipe a pu ainsi identifier, sur une population de plus de 2000 personnes et 8000 échantillons issus de différents tissus, 393 séquences présentes dans les îlots CpG capables de prédire l’âge biologique des patients. Ces marqueurs ont le double intérêt d’être hautement sensibles (une prédiction fiable à plus de 96%) mais aussi très spécifiques des tissus dans lesquels ils sont retrouvés [13]. Plus récemment, une étude effectuée uniquement sur le sang a permis de prédire l’âge biologique à partir de seulement trois sites CpG spécifiques, démontrant ainsi que la théorie de « l’horloge épigénétique » est belle est bien réelle [14].
La voie de l’avenir : les « épimédicaments »
Partant du principe que les modifications épigénétiques sont généralement réversibles, beaucoup d’espoirs se tournent aujourd’hui vers des thérapies ciblées pouvant corriger ces changements: cette idée conduit au développement d’epimédicaments, capables d’agir pour éliminer les modifications anormales de notre ADN. Deux familles de molécules ont été d’ores et déjà été développées : les inhibiteurs méthylation de l’ADN (DNMTi) et les médicaments ciblant la modification des histones (HDACi).
Malheureusement, à l’heure actuelle, ces médicaments ne sont pas assez spécifique pour avoir une action ciblée et sans effets secondaires. De nombreuses équipes de recherches travaillent néanmoins sur l’amélioration des molécules existantes voire sur la mise en place de multi-thérapies associant épimédicaments et molécules classiques.
Partie 1 : Mesure du vieillissement : La Cartographie PhysiologiqueⓇ
 Cette méthode de métrologie du vieillissement et de l’âge physiologique procède au bilan de diverses caractéristiques biologiques et physiques qui ont un lien avéré avec les processus de vieillissement (dosage du cholestérol, appui monopodal, obésité abdominale…).
Cette méthode de métrologie du vieillissement et de l’âge physiologique procède au bilan de diverses caractéristiques biologiques et physiques qui ont un lien avéré avec les processus de vieillissement (dosage du cholestérol, appui monopodal, obésité abdominale…).
Partie 2 : Mesure du vieillissement : Les 18 biomarqueurs de l’Université de Duke
 Cette méthode suit le déclin de plusieurs systèmes organiques (cardiovasculaire, pulmonaire, rénal, hépatique, parodontal, immunitaire) à travers la quantification de 18 marqueurs de maladies chroniques relatives à l’âge. Cette technique a été mise au point par une équipe de recherche de l’Université de Duke, supervisée par le Dr. Daniel Belsky.
Cette méthode suit le déclin de plusieurs systèmes organiques (cardiovasculaire, pulmonaire, rénal, hépatique, parodontal, immunitaire) à travers la quantification de 18 marqueurs de maladies chroniques relatives à l’âge. Cette technique a été mise au point par une équipe de recherche de l’Université de Duke, supervisée par le Dr. Daniel Belsky.
Partie 4 : Les télomères, témoins de l’âge physiologique, un outil pour la mesure du vieillissement
 Les télomères, qui sont les extrémités de nos chromosomes, voient leur longueur raccourcir au cours du vieillissement. On compte plusieurs techniques permettant de mesurer leur longueur (TRFs, Q-Fish, PCR-Q, STELA…) basées sur les principes de Southern Blot, la PCR et l’hybridation in situ.
Les télomères, qui sont les extrémités de nos chromosomes, voient leur longueur raccourcir au cours du vieillissement. On compte plusieurs techniques permettant de mesurer leur longueur (TRFs, Q-Fish, PCR-Q, STELA…) basées sur les principes de Southern Blot, la PCR et l’hybridation in situ.
Références
[1] Jones MJ, Goodman SJ, Kobor MS. DNA methylation and healthy human aging. Aging Cell. 2015;14(6):924-932
[2] Weisenberger DJ. Characterizing DNA methylation alterations from The Cancer Genome Atlas. The Journal of Clinical Investigation. 2014;124(1):17-23
[3] Wüllner U, Kaut O, deBoni L, Piston D, Schmitt I. DNA methylation in Parkinson’s disease. J Neurochem. 2016 Oct;139 Suppl 1:108-120
[4] Horvath S, Zhang Y, Langfelder P, et al. Aging effects on DNA methylation modules in human brain and blood tissue. Genome Biology. 2012;13(10):R97
[5] Xu X. DNA methylation and cognitive aging. Oncotarget. 2015;6(16):13922-13932.
[6] Chambers JC, Loh M, Lehne B, et al. Epigenome-wide association of DNA methylation markers in peripheral blood from Indian Asians and Europeans with incident type 2 diabetes: a nested case-control study. The lancet Diabetes & endocrinology. 2015;3(7):526-534
[7] Johnson AA, Akman K, Calimport SRG, Wuttke D, Stolzing A, de Magalhães JP. The Role of DNA Methylation in Aging, Rejuvenation, and Age-Related Disease. Rejuvenation Research. 2012;15(5):483-494
[8] Zampieri M., Ciccarone F., Calabrese R. , Franceschi C., Burkle A., Caiafa P., Reconfiguration of DNA methylation in aging. Mech. of Ageing and Dev., 2015, vol 151; P. 60-70
[9] Ehrlich M, Gama-Sosa MA, Huang L-H, Midgett RM, Kuo KC, McCune RA, Gehrke C. Amount and distribution of 5-methylcytosine in human DNA from different types of tissues or cells. Nucleic Acids Res. 1982;10:2709–21.
[10] Jung M, Pfeifer GP. Aging and DNA methylation. BMC Biology. 2015;13:7
[11] Wilson VL, Smith RA, Ma S, Cutler RG. Genomic 5-methyldeoxycytidine decreases with age. J Biol Chem. 1987;262:9948–51
[12] Wilson VL, Jones PA. DNA methylation decreases in aging but not in immortal cells. Science. 1983;220:1055–7
[13] Horvath S. DNA methylation age of human tissues and cell types. Genome Biol. 2013;14:R115
[14] Weidner CI, Lin Q, Koch CM, Eisele L, Beier F, Ziegler P, et al. Aging of blood can be tracked by DNA methylation changes at just three CpG sites. Genome Biol. 2014;15:R24
Dr. Marion Tible

Author/Reviewer
Auteure/Relectrice
Marion Tible has a PhD in cellular biology and physiopathology. Formerly a researcher in thematics varying from cardiology to neurodegenerative diseases, she is now part of Long Long Life team and is involved in scientific writing and anti-aging research.
More about the Long Long Life team
Marion Tible est docteur en biologie cellulaire et physiopathologie. Ancienne chercheuse dans des thématiques oscillant de la cardiologie aux maladies neurodégénératives, elle est aujourd’hui impliquée au sein de Long Long Life pour la rédaction scientifique et la recherche contre le vieillissement.
En savoir plus sur l’équipe de Long Long Life
Dr Guilhem Velvé Casquillas

Author/Reviewer
Auteur/Relecteur
Physics PhD, CEO NBIC Valley, CEO Long Long Life, CEO Elvesys Microfluidic Innovation Center
More about the Long Long Life team
Docteur en physique, CEO NBIC Valley, CEO Long Long Life, CEO Elvesys Microfluidic Innovation Center
En savoir plus sur l’équipe de Long Long Life